 Tenk på ett gen
Tenk på ett gen
(Fritt utvalg fra Darwins Doubt; S.C.Meyer, Harper One, 2013, Ch.11) {med oversetters kommentar i klamme-parentes}
Da Stephen C. Meyer publiserte en vitenskapelig artikkel om den kambriske eksplosjonen, og problemene den reiste med forklaringsbehov for informasjonen som plutselig dukket opp i kambriske dyr, argumenterte han for at så mye ny informasjon vanligvis skyldes en intelligens. Det voldte stor oppsikt, og endte med at redaktøren av magasinet, Richard Steinberg, som ga ut artikkelen ble presset ut av stillingen. Til tross for dette, tok det tid før det kom noe vitenskapelig svar på utfordringen Meyer hadde reist i sin artikkel. Til slutt kom det opp svar av folk tilknyttet National Center for Science Education. I en polemisk formet artikkel, hevdet de at problemet med opprinnelsen til genetisk informasjon var løst. ("The Origin of New Genes: Glimpses from the young and old" (Nature Reviews Genetics i 2003). Det ble hevdet at denne artikkelen, der M. Long -en evolusjonsbiolog ved Chicago universitet som én forfatter, var representativ for omfattende "vitenskapelig litteratur som dokumenter opprinnelsen til nye gener". (7) Ett annet sted har Matzke sammen med biolog Paul Gross, erklært at artikkelen av Long "går gjennom alle mutasjons-prosesser involvert i opprinnelsen til nye gener og lister så opp dusinvis av eks. der forskningsgrupper har rekonstruert genenes opphav" (9). Ifølge deres syn "vet kompetente vitenskapsfolk hvordan ny genetisk informasjon oppstår." (10)
Men vet virkelig evolusjonære biologer dette? Om vi tar en nærmere kikk på det artikkelen angivelig skal vise (11), er det ikke vanskelig å spore svakheter.
Bilde 1. Meyers alternativ høstet storm
Det var en gang ett gen
Long-artikkelen peker på flere studier som gir seg ut for å forklare evolusjon av ulike gen. Denne type studier begynner gjerne med å ta ett gen, og så søke til en finner andre lignende (homologe) gen med dette. Så søker en å spore historien til litt ulike tilsvarende gener, bakover mot en hypotetisk felles 'stam-gen'. En søker gjerne gjennom databaser med gensekvenser, og ser etter lignende sekvenser i ulike taksonomiske grupper, ofte i nært beslektede arter. så går en videre med å foreslå evolusjonære scenarier der ett 'stam-gen' dupliserer seg selv (12), og så duplikatet og originalen utvikler seg ulikt, som følge av påfølgende mutasjoner i hvert gen.
De neste scenariene inneholder gjerne ulike slag mutasjoner, innsettinger, dupliseringer, forflytninger, retroposisjonering, sideveis gen-overføring i tillegg til naturlig seleksjon. Evolusjonsbiologene som utfører slike studier postulerer at moderne gener oppsto som resultat av disse ulike mutasjons-prosessene gjennom lang tid. Siden informasjonen i moderne gener formodentlig er annerledes enn i hypotetiske 'stam-gener', betraktes mutasjonsprosessene som ansvarlige for disse ulikhetene, og forklaringen på opprinnelsen til genetisk informasjon.
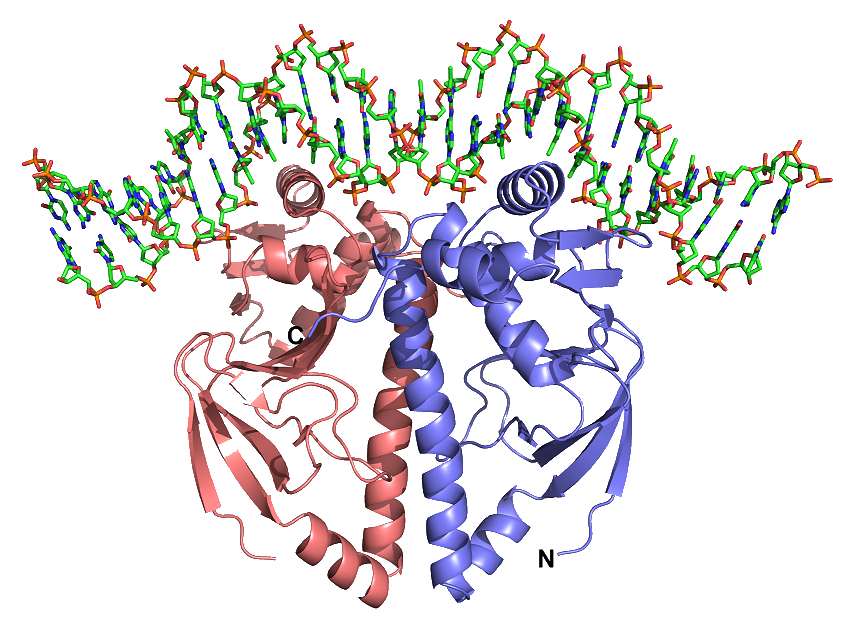 Ved nærmere undersøkelse viser ingen av disse artiklene hvordan mutasjoner og naturlig seleksjon, virkelig kunne finne nye gener, eller proteiner med 3-dimensjonal folding -i første omgang. Heller ikke viser de hvordan det kunne skje på tilgjengelig tid, verken i livets historie eller i forhold til kambrium. Artiklene formoder eksistensen av betydelige pre-eksisterende genetisk informasjon, og foreslår så ulike mekanismer som sakte kunne ha utviklet, eller slått sammen disse genene til større sammensetninger. I beste fall sporer disse artiklene historien til pre-eksistente gener, heller enn å forklare opphavet til de opprinnelige genene.
Ved nærmere undersøkelse viser ingen av disse artiklene hvordan mutasjoner og naturlig seleksjon, virkelig kunne finne nye gener, eller proteiner med 3-dimensjonal folding -i første omgang. Heller ikke viser de hvordan det kunne skje på tilgjengelig tid, verken i livets historie eller i forhold til kambrium. Artiklene formoder eksistensen av betydelige pre-eksisterende genetisk informasjon, og foreslår så ulike mekanismer som sakte kunne ha utviklet, eller slått sammen disse genene til større sammensetninger. I beste fall sporer disse artiklene historien til pre-eksistente gener, heller enn å forklare opphavet til de opprinnelige genene.
Bilde 2. Proteiner må ha rett 3-dimensjonal folding for å fungere
Denne slags scenarier kan foreslå potensielt fruktbare forsknings-veier. Men å holde for at de demonstrerer faktum, eller er en fullgod forklaring, blir feilaktig. Ingen av scenariene i Long-artikkelen viser matematisk sannsynlighet eller eksperimentell troverdighet til mutasjons-mekanismene, som de selv hevder er forklaring til opprinnelsen av genene. Heller ikke observerer de aktuelle mutasjons-mekanismer i aksjon. I beste fall gir de rekonstruksjoner av noen få begivenheter, ut fra en sekvens av mange formodede. Genet de starter med, formodes å ha eksistert på basis av likhet med to eller flere eksisterende gener, som er de eneste observerte enheter som disse omhyggelig utarbeidede scenariene er basert på.
At scenariene slik sett er basert på ulike forutsetninger og postulater, diskvalifiserer dem ikke fra å bli tatt i betraktning. Men om de fullgodt forklarer opprinnelsen til genetisk informasjon, avhenger av hvorvidt eksistensen av entitetene de slutter tilbake til, og troverdigheten til de mutasjonelle mekanismene, som de postulerer. La oss først se på:
Felles stam-gen?
Nesten alle scenariene i Long-artikkelen, starter med et felles opphavs-gen, som to eller flere mer moderne gener gikk ut fra og utviklet seg. Scenariene oppfatter likhet i gensekvens som utvetydig bevis på felles opphav, slik en forutsetter i fylogenetiske rekonstruksjoner. Men dette stemmer ikke alltid. i) Noen ganger kan genetisk likhet inntreffe mellom arter som ikke kan bli forklart ved arv fra en felles stamfar, og ii) det er faktisk andre mulige forklaringer.
 i) Like gensekvenser kan ha utviklet seg uavhengig i to parallelle utviklings-linjer, som starter med to ulike gener ('konvergent evolusjon'). Det er mange slike eks. nå (13). F.eks. benytter både flaggermus og hvaler, samme type ekkolokalisering, inklusive lignende gener og proteiner for dette. Dette har fått evolusjonsbiologer til å foreslå at de begge stammer fra en felles stamfar, som ikke opprinnelig hadde dette systemet. (14)
i) Like gensekvenser kan ha utviklet seg uavhengig i to parallelle utviklings-linjer, som starter med to ulike gener ('konvergent evolusjon'). Det er mange slike eks. nå (13). F.eks. benytter både flaggermus og hvaler, samme type ekkolokalisering, inklusive lignende gener og proteiner for dette. Dette har fått evolusjonsbiologer til å foreslå at de begge stammer fra en felles stamfar, som ikke opprinnelig hadde dette systemet. (14)
ii) I tillegg er det prinsipielt mulig at liknende gener er separat designet, til å møte lignende funksjonelle behov i ulike organisme-kontekster. Dette kommer vi nærmere tilbake til i Kap.17-20 (lenke). Poenget med å nevne det her, er bare for å demonstrere at det ikke nødvendigvis forutsetter felles stamfar.
Bilde 3. Genduplisering + mutasjoner
 Orfan gener
Orfan gener
Noen gener og informasjonsrike sekvenser som de inneholder, kan sikkert ikke bli forklart ved å henvise til den slags scenarier Long-artikkelen gjør. Alle de forsøker å forklare ved hjelp av nedstamning ved modifikasjoner, fra felles stamgener. Men genomstudier viser nå hundre-tusenvis av gener i mange ulike organismer, som ikke viser noen betydelig sekvens-likhet med noen andre kjente gener (15). Disse taksonomisk begrensede genene, eller 'orfan -gener' for ('Open Reading FrAmes of uNknown origin.'), dekker nå det fylogenetiske landskapet. De har dukket opp i alle hovedgrupper av organismer, dyr og planter, organismer både med (eukaryote) kjerne og uten (prokaryote). I noen organismer er så mye som halvparten av genomet bestående av orfan-gener. (16) {Typisk andel er ca. 10-30% -oversetters tilføyelse.} Disse anerkjennes nå av stadig bredere lag av biologer, som gjerne forsøker å (bort)forklare disse med at deres opprinnelse er ut av intet (de novo). Noen vil sikkert poengtere at ettersom flere og flere genom sekvensieres, så vil det dukke opp gener med likhet til disse orfan-genene. Men så langt har tendensen vært motsatt, man har oppdaget flere og flere orfan-gener, uten å finne noe slikt som korresponderende homologe (likeartede) gener. Trenden med et økende antall orfan-gener fortsetter å vokse. (17)
Bilde 4. Bok om orfan gener
Troverdigheten ved mutasjons-prosessene
Selv om evolusjonsbiologer kunne etablere eksistensen av ett 'felles opprinnelig gen', som oppstarten på sine scenarier, ville ikke det bevise tilforlateligheten ved en neo-darwinistisk mekanisme for å samle opp genetisk informasjon fra den 'stamfaren'. Termen 'plausibilitet' i denne sammenheng, har en vitenskapelig og metodologisk signifikant betydning. Studier i vitenskapsfilosofi viser at suksessfylte forklaringer i historiske vitenskaper trenger å forsyne 'kausalt tilstrekkelige' forklaringer. Det innebærer å forklaringer som siterer en årsak eller mekanisme, i stand til å produsere effekten det er snakk om. I 'Artenes opprinnelse' forsøkte Darwin gjentatte ganger å vise at hans teori tilfredsstilte dette kriteriet, som da ble kalt 'vera causa' eller 'sann årsaks' kriterium. I kap. 3 forsøkte han f.eks. å demonstrere kausal tilstrekkelighet for naturlig seleksjon, ved å trekke analogier mellom det og styrken ved dyre-oppdrett, og ved å ekstrapolere fra observerte tilfeller av små-skala evolusjonære forandringer -over korte tidsperioder.
 På dette viset knyttet Darwin til vitenskaps-prinsippet som en av hans vitenskapelige rolle-modeller, geologen Charlew Lyell, benyttet. Lyell insisterte på at gode forklaringer for opprinnelsen til geologiske trekk, skulle siterer 'årsaker som er i virksomhet nå, -årsaker som er kjent fra nåværende erfaring å ha kapasitet til å framskaffe den søkte effekten' (18). Hvordan er det med scenariene benyttet i Longs artikkel: tilfredsstiller de disse kriteriene? Dupliserings mutasjoner og andre typer tilfeldige mutasjoner, er sammen med naturlig seleksjon, klart årsaker som er i virksomhet nå. Men har disse prosessene demonstrert evnen til å produsere hva det spørres etter, nemlig ny genetisk informasjon som er nødvendig for strukturelle innovasjoner i livets historie? Det er flere gode grunnet til å mene at de ikke har gjort det.
På dette viset knyttet Darwin til vitenskaps-prinsippet som en av hans vitenskapelige rolle-modeller, geologen Charlew Lyell, benyttet. Lyell insisterte på at gode forklaringer for opprinnelsen til geologiske trekk, skulle siterer 'årsaker som er i virksomhet nå, -årsaker som er kjent fra nåværende erfaring å ha kapasitet til å framskaffe den søkte effekten' (18). Hvordan er det med scenariene benyttet i Longs artikkel: tilfredsstiller de disse kriteriene? Dupliserings mutasjoner og andre typer tilfeldige mutasjoner, er sammen med naturlig seleksjon, klart årsaker som er i virksomhet nå. Men har disse prosessene demonstrert evnen til å produsere hva det spørres etter, nemlig ny genetisk informasjon som er nødvendig for strukturelle innovasjoner i livets historie? Det er flere gode grunnet til å mene at de ikke har gjort det.
Å tigge spørsmål
De fleste av mutasjons-scenariene som evolusjons-biologer foreslår i Long artikkelen: innsettinger, dupliseringer, forflytninger, retroposisjonering, sideveis gen-overføring -forutsetter betydelige mengder av pre-eksisterende genetisk informasjon. Det er kun 'de novo' dannelse, som ikke starter med det. Denne pre-eksisterende, funksjonelt spesifiserte informasjon er i noen tilfeller tilstrekkelig til å kode for ett helt protein, eller spesifikk protein-fold. Scenariene bare forutsetter, uten å forsøke å forklare hvordan noen av mekanismene de anviser til kan ha løst det kombinatoriske søke-problemet vi tok opp i forrige kapittel
Bilde 5. Ulike måter gener modifiseres
{Ekskurs eksoner og introner: Genene hos organismer med cellekjerne er organisert i to slags DNA-segmenter, eksoner og introner. Et ekson koder for en serie aminosyrer som utgjør et avsnitt av det proteinet genet bestemmer. Mellom to eksoner sitter et intron, som ikke koder for noen aminosyrer. Enkelte mener at introner er med på å styre genenes virksomhet på en måte som foreløpig ikke er forstått. Når genet blir avlest, produseres et RNA-molekyl som representerer hele genet, både alle eksoner og alle introner. Men før proteinsyntesen finner sted, blir alle RNA-stykker som svarer til intronene kuttet ut, slik at bare eksonene er representert i det RNA som styrer proteinsyntesen på ribosomene. Fra: snl eksoner -organisering}
Tilhengere av ekson-forflytninger (shuffling) anviser modulære seksjoner av genomet, som tilfeldig arrangerer eller rearrangere seg selv, til helt nye gener. Ikke ulikt om en setter sammen en helt ny artikkel, ut fra avsnitt fra tidligere artikler. Ett enkelt ekson kan kode for en viktig del av en protein-struktur, slik som en funksjonell protein-folding. Dette er et faktum som tilhengerne tar med i sine forsøk på å forklare nye proteiner. De forutsetter at eksoner blindt kan 'skuffes omkring' og sette sammen gener. Men det viser seg at denne mekanismen ikke kan danne nye protein-foldinger, som behøves for å danne nye kropps-former. Enten er eksonet stort nok til at det allerede koder for en protein-folding, eller så må flere eksoner kombineres for å danne en stabil protein-fold. I det siste tilfellet, dukker det opp f.eks. problemet som kalles motsatt side-kjede interaksjon, som vi skal se vil hindre protein-fold dannelse. Sideveis gen-overføring innebærer å overføre ett pre-eksisterende gen (vanligvis en bakterie), inn i en annens organismes genom. Overføring av mobile genetiske elementer inntreffer likedan når pre-eksistente gener innelukket i sirkulære DNA-sammenføyninger, kalt plasmider-kommer fra en organisme inn i en annen, og til slutt inkluderes i genomet. Såkalte transposoner (hoppende gener) kan forflytte seg fra én plass i genomet til en annen (19). Gen-fusjon inntreffer når to motsvarende pre-eksisterende gener, linker seg sammen, etter sletting av mellomliggende genetisk materiale (20).
Hver av disse mutasjons-mekanismene forutsetter pre-eksistente gener, og noen av dem forutsetter også sofistikerte pre-eksistente molekylære maskiner, (f.eks. reversed transcritase enzyme). Slike molekylære maskiner krever andre kilder av genetisk materiale, som klart gir grunnlag for å spørre hvor de kom fra. De mekanismene som nevnes kan sammenliknes med å forsøke å produsere en ny bok, ved å kopiere og redigere sidene fra eksisterende bøker, og så tilfeldig rearrangere de nye sidene. Det synes klart at slike tilfeldige omarrangeringer ikke vil ha stor sjanse for å produsere noe litterært mesterverk, enn si gi sammenhengende lesning. Det er også stor forskjell på å å flytte, eller lett moderere pre-eksisterende sekvens-spesifikke moduler av funksjonell informasjon, og å forklare hvordan disse modulene ble til i første omgang.
 Evolusjon av intet ('ex nihilo')?
Evolusjon av intet ('ex nihilo')?
Long siterer minst en type mutasjon som ikke forutsetter pre-eksisterende genetisk informasjon. F.eks. var det en artikkel han beskrev, som fant ut at en 'uvanlig regulerende region' ikke utviklet seg. I stedet ble den plutselig til: "Den var opprinnelig, dannet nytt av tilfeldig sammenstilling av egnede sekvenser (21). Flere andre artikler kaller på 'de-novo' opprinnelse. F.eks. nevner Long en studie som forsøker å forklare opprinnelsen til ett 'anti-frys' protein i en Antarktisk fisk. Her nevnes 'de-novo' forstørrelse av en kort DNA-sekvens, for å danne ett nytt protein med en ny funksjon" (22). Long siterer en artikkel i Science for å forklare opprinnelsen til 'de-novo' dannelse av enkelte gen-segmenter som koder for protein-domener. Her dukket ett ekson plutselig fram fra en unik ikke-protein kodende sekvens". (23) En artikkel fra 2009 refererte til minst 3 de-novo dannelser av menneskelig protein-kodende gener, siden atskillelsen fra sjimpanser". I det tilfellet har ingen av dem "noen protein-kodende homologe gener i noe annet genom" (24). En enda nyere artikkel fra PLoS Genetics rapporterte om "60 nye protein-kodende gener som oppsto 'de novo' på den menneskelige linjen, siden atskillelse med sjimpanser" (25). Det var et funn som ble kalt mye større enn ett tidligere, et konservativt estimat" (26).
Bilde 6. Ett gen 'dukker plutselig opp'
En annen artikkel fra 2009 i journalen 'Genome Research' ble passende kalt "Darwinistisk alkymi": "Menneskelige gener fra ikke-protein kodende RNA". Oppdukkingen av komplette, funksjonelle gener, orfan-gener og funksjonelle proteiner fra 'junk-DNA' ville synes høyst usannsynlig.." (27) Artikkelen påsto likevel, uten å grunngi, at "evolusjon ved naturlig seleksjon, kan danne nye funksjonelle elementer fra tilsynelatende ikke-funksjonell DNA". Det kan sammenlignes med 'prosessen der molekylære evolusjonister omdanner bly til gull' (28). Nærværet av unike gen-sekvenser kan tvinge forskere til å 'kalle på' de novo opprinnelse av gener oftere enn de ønsker. Etter en studie av bananfluer som rapporterte at 12% av nylig oppdukkede gener i Drosophila malangaster undergruppen, kan ha oppstått 'de novo' fra ikke-protein kodende DNA." (29) Forfatteren fortsatte her med å erkjenne at å kalle på denne mekanismen stilte ett alvorlig problem for evolusjonsteori, siden det ikke forklarte opprinnelsen til noen av dets 'ikke-trivielle krav for funksjonalitet." (30)
 Forfatteren forutsetter at pre-adapsjon kan ha spilt en rolle. Men han tilføyer ingen ytterligere forklaring, siden det bare innebærer når (før seleksjon spilte en rolle) og hvor (ikke-protein kodende DNA), ikke hvordan de aktuelle genene oppsto. Evolusjonære biologer benytter typisk termen 'de novo opprinnelse' for å ha noe å henføre til, når de ikke kan forklare økning i genetisk informasjon. Det henfører ikke til noen kjent mutasjons-prosess. Så enten 'tigger' forklaringene spørsmål om opprinnelig informasjon, eller så foretar de uforklarlige hopp, evolusjonær skapelse 'ex nihilo'. Dermed forklarer ikke scenariene i Longs artikkel opprinnelsen til funksjonell spesifisert informasjon, verken i gener eller seksjoner av gener. Det ville kreve en årsak som kunne løse det kombinatoriske-søke spørsmålet i forrige kapittel. Men ingen av artiklene i Longs artikkel, verken adresserer eller demonstrerer matematisk troverdighet i mekanismene de henviser til. En kan ikke løse et problem eller imøtegå ett argument ved å unnlate å adressere det. (31) {Vi har tidligere presentert en ikke-tilfeldig evolusjonær hypotese i slike situasjoner.}
Forfatteren forutsetter at pre-adapsjon kan ha spilt en rolle. Men han tilføyer ingen ytterligere forklaring, siden det bare innebærer når (før seleksjon spilte en rolle) og hvor (ikke-protein kodende DNA), ikke hvordan de aktuelle genene oppsto. Evolusjonære biologer benytter typisk termen 'de novo opprinnelse' for å ha noe å henføre til, når de ikke kan forklare økning i genetisk informasjon. Det henfører ikke til noen kjent mutasjons-prosess. Så enten 'tigger' forklaringene spørsmål om opprinnelig informasjon, eller så foretar de uforklarlige hopp, evolusjonær skapelse 'ex nihilo'. Dermed forklarer ikke scenariene i Longs artikkel opprinnelsen til funksjonell spesifisert informasjon, verken i gener eller seksjoner av gener. Det ville kreve en årsak som kunne løse det kombinatoriske-søke spørsmålet i forrige kapittel. Men ingen av artiklene i Longs artikkel, verken adresserer eller demonstrerer matematisk troverdighet i mekanismene de henviser til. En kan ikke løse et problem eller imøtegå ett argument ved å unnlate å adressere det. (31) {Vi har tidligere presentert en ikke-tilfeldig evolusjonær hypotese i slike situasjoner.}
Bilde 7. Sannsynlighet for å finne fugnerende proteiner (fra kap.10)
Protein-folding:
troverdige, men irrelevante scenarier
I nær sammenheng med ovenfor nevnte svakheter i Longs artikkel, knyttet til opprinnelse av nye gener, så forsøker en heller ikke å forklare hvordan nye gener, som er tilstrekkelig ulik hverandre til å danne nye 3-dimensjonale protein-foldinger. Som vi så i kap. 10, er nye protein-foldinger den minste av valgbare, strukturelle innovasjoner, og mye større strukturelle innovasjoner i historien, er avhengig av dem. I stedet prøver Long vanligvis å forklare opprinnelsen til homologe gener, som produserer samme foldingsstruktur, som utfører samme, eller en nært tilknyttet, funksjonalitet. F.eks. sammenligner han to gener, RNASE1 og RNASE1B, som koder for homologe fordøyelses enzymer (32). De to proteinene utøver tilnærmet samme funksjonalitet, og har 93% likhet i DNA-strukturen. Dermed vil biologer forvente at de benytter samme protein-folding, for å utføre sine nært beslektede oppgaver. Likedan refereres ett gen som koder for et histon-protein, Cid, i to nært relaterte arter av bananfluer. Studien prøvde ikke å finne opprinnelsen til genet. Den bare sammenlignet de to litt ulike proteinene, hvorav 17 aminosyrer var endret i en sekvens av 226. En så liten endring er ikke sannsynlig at oversettes i ulike protein-foldinger. Dersom naturlige foldinger er noe i nærheten av så liten forskjell (7,5%), har de ikke ulike foldinger. Long siterer også to studier av FOXP2, ett gen involvert i å regulere gen-uttrykk hos sjimpanser og mennesker. Men proteinet som ble kodet for av dette genet, trengte bare en endring på 2 aminosyrer på den menneskelige linjen, i forhold til sjimpanser. {I det hele er proteiner såpass 'fast oppsatt' at det ikke er her de store endringer mellom artene forekommer- oversetters tilføyelse.}
 Å forklare opphavet til strukturell innovasjon, innebærer mer enn bare å sammenligne ulike versjoner av samme gener, og forsøke spore et opphav til disse endringene. Det innebærer å finne forklaring til nytt genetisk materiale, som danner nye tredimensjonale protein-foldinger, slik at større strukturelle innovasjoner kan inntreffe. Så selv om disse scenariene i og for seg er greie nok, er de ikke relevante for å forklare opprinnelse til ny strukturell innovasjon, som forekommer i kambrium-perioden, -eller i noen andre begivenheter i livets historie.
Å forklare opphavet til strukturell innovasjon, innebærer mer enn bare å sammenligne ulike versjoner av samme gener, og forsøke spore et opphav til disse endringene. Det innebærer å finne forklaring til nytt genetisk materiale, som danner nye tredimensjonale protein-foldinger, slik at større strukturelle innovasjoner kan inntreffe. Så selv om disse scenariene i og for seg er greie nok, er de ikke relevante for å forklare opprinnelse til ny strukturell innovasjon, som forekommer i kambrium-perioden, -eller i noen andre begivenheter i livets historie.
Relevante, men ikke-troverdige scenarier
I noen få tilfeller er de evolusjonære scenariene i Longs artikkel, forsøk på å forklare gener som er tilstrekkelig ulike til at de kunne kode for proteiner med ulik folding. F.eks. diskuterer Long flere artikler som sammenligner ekson-skyfling med flytting av protein-domener. Ett protein-domene er en stabil, tredimensjonal proteinstruktur eller folding, som er sammensatt av mange mindre eller 'sekundære' strukturer. Mange komplekse proteiner har tallrike domener, som hver foreviser en unik folding eller tre-dimensjonal struktur. En versjon av hypotesen om ekson-skyfling, impliserer at hvert ekson koder for ett spesifikt protein domene. Det visualiserer tilfeldig kutting og spleising, og utøver flyttinger og rekombinasjoner av gener. Det resulterende sammensatte genet, vil så kode for en nytt sammensatt protein-struktur. Som Longe forslår: "Ekson-skyfling, som også er kjent som domene-skyfling, rekombinerer ofte sekvenser som koder for ulike protein-domener, for å skape mosaikk-proteiner"(36). Selv om dette kanskje er det mest relevante scenariet, så er det likevel problematisk av flere grunner:
Bilde 8. Tilfeldige forflytninger forklarer ikke
i) For en vitenskapsmann som jobber med proteiner, så er ett protein-domene ekvivalent med en protein-fold, selv om distinkte protein strukturer (foldinger), kan være sammensatt av flere mindre domener eller foldinger. Så hypotesen om ekson-skyfling forutsetter tidligere eksistens av en betydelig mengde genetisk informasjon, tilstrekkelig til å danne ett protein-domene eller protein-folding. Nå benyttes også termen 'protein-domene' litt annerledes av noen, som domener med mindre strukturelle enheter, slik som fragmenter av en fold som er laget av atskillige enheter av sekundære strukturer (f.eks. alfa-helikser og beta-strands (eng). Ekson-skyfling i denne terminologien ville innebære konstruksjon av ny protein-struktur, ved å kombinere disse mindre fragmentene. Men i de fleste tilfeller, når amino-syre kjeden i ett domene, blir delt i fragmenter, så vil de resulterende isolerte stykkene opphøre å beholde sine opprinnelige former. {Helheten er større enn summen av delene -oversetters kommentar}. Og selv om slik skyfling av del-strukturer var mulig, så ville den måtte forutsette funksjonell informasjon som ikke er forklart. Det gjelder både informasjon til å spesifisere de mindre elementene, men også påkrevd informasjon til å arrangere disse mindre enhetene til stabile foldinger, og endelig funksjonelle proteiner.
ii) Så bygger også scenariet på en urealistisk forutsetning: at ekson-omgivelsene korresponderer med grenseområdet til ett protein-domenet eller tilsvarende folding. Men i eksisterende gener så gjelder ikke dette (38). Om hypotesen med skyfling av eksoner hadde forklart hvordan aktuelle proteiner hadde kommet til, så kunne det ha vært samsvar mellom nevnte grenseområder. Men fraværet av et slikt samsvar, indikerer at ekson-skyfling ikke gjør rede for opprinnelsen til kjente, sammensatte protein-strukturer.
 iii) En tredje mangel på samsvar finner vi i begrepet 'side-kjeder' (side chains). Alle de 20 protein-formende aminosyrene har sitt eget rammeverk (bakcbone-'ryggrad'), som kan kalles for en 'side-kjede'. Disse stikker ut i nesten rett vinkel fra rammeverket. Samhandlinger mellom slike 'side-kjeder' avgjør hvorvidt det vil bygges opp større, stabile 3-dimensjonale foldinger (39). Siden elementene i mindre, sekundære protein-enheter er omgitt av 'side-kjeder', så kan de ikke kombineres om ikke aktuelle side-kjeder er komplementære. Selv trente vitenskapsfolk, som har jobbet med proteiner i årevis, kan ha problemer med å designe sekvenser som kan gi stabile protein-foldinger (42). Manglende interaksjon fra side-kjedene (43) forhindrer det. Som molekylær-biolog Ann Gauger uttrykte det: " Således er alpha-helikser og 'beta-svøp' sekvens-avhengige strukturelle elementer av protein-foldinger. En kan ikke bytte dem rundt, som med Lego-brikker." (44) {Forskjellen er at mens Lego-fabrikanten driver utstrakt hemmelighold, over noe som relativt enkelt kan forstås, så har 'livets fabrikant' lagt koden åpent fram, men selv de skarpeste hoder har problemer med å forstå den -oversetters kommentar.}
iii) En tredje mangel på samsvar finner vi i begrepet 'side-kjeder' (side chains). Alle de 20 protein-formende aminosyrene har sitt eget rammeverk (bakcbone-'ryggrad'), som kan kalles for en 'side-kjede'. Disse stikker ut i nesten rett vinkel fra rammeverket. Samhandlinger mellom slike 'side-kjeder' avgjør hvorvidt det vil bygges opp større, stabile 3-dimensjonale foldinger (39). Siden elementene i mindre, sekundære protein-enheter er omgitt av 'side-kjeder', så kan de ikke kombineres om ikke aktuelle side-kjeder er komplementære. Selv trente vitenskapsfolk, som har jobbet med proteiner i årevis, kan ha problemer med å designe sekvenser som kan gi stabile protein-foldinger (42). Manglende interaksjon fra side-kjedene (43) forhindrer det. Som molekylær-biolog Ann Gauger uttrykte det: " Således er alpha-helikser og 'beta-svøp' sekvens-avhengige strukturelle elementer av protein-foldinger. En kan ikke bytte dem rundt, som med Lego-brikker." (44) {Forskjellen er at mens Lego-fabrikanten driver utstrakt hemmelighold, over noe som relativt enkelt kan forstås, så har 'livets fabrikant' lagt koden åpent fram, men selv de skarpeste hoder har problemer med å forstå den -oversetters kommentar.}
Bilde 9. Slik kan det føles for noen-både her og der(?)
Selv innen de minste celler, er det mange misjons-kritiske funksjoner som krever minst 150 aminosyre-kjeder, eller mange protein-foldinger. Så slike problem kommer en mange ganger ikke unna, i løpet av livets historie. Poenget er ikke så mye om dette problemet har blitt løst (-for det har det jo), som hvorvidt neo-darwinistiske mekanismer som liter på tilfeldige mutasjoner, i form av tilfeldig skyfling av eksoner, skulle være en troverdig forklaring. Selv om tilhengere av ekson-skyfling ikke utfordrer Axe sitt kvantitative estimat, som er eksperimentelt oppnådd, så påstås det selvsikkert fra disse at "ekson-skyfling ofte rekombinerer sekvenser som koder for ulike proteiner, til å danne mosaikk-proteiner." {En hevder altså å ha løst problemet, uten å kommentere vanskeligheter knyttet til det. At det i tillegg produseres mye bla, bla, som ødelegger tillit til ordenes betydning, dropper jeg. I det hele står det mye mer i boka enn det jeg har valgt å ta med, så jeg oppfordrer engelsk-kyndige til å lese den.}
Referanser:
9. Matzke og Gross, "Analyzing Critical Analysis," 42
10.
Matzke og Gross, "Analyzing Critical Analysis," 42
11. Matzke og Gross, "Analyzing Critical Analysis," 42
13. F.eks. Zhen et al, "Parallell Molecular Evolution in an Herbivore Community"; Li et al, "The Hearing Gene Prestin Unites Echolocating Bats and Whales"
14. Shen et al, "Parallel Evolution of Auditory Genes for Echolocalisation in Bats and Toothed Whales"; Jones, "Molecular Evolution."
15. Khalturin et al, "More Than just Orphans"; "Rhizome of Life, Catastrophes, Sequence Exchanges, Gene Creations, and Giant virures";
16. Suen et al, "The Genome Sequence of the Leaf-Cutter Ant Attta cephalotes Reveals Insights int its Obligate Symbolic Lifestyle" ("We also found 9.361 proteins that are unique to A. cephalotes, representing over half of its predicted proteome,"5; Smith et al, "Draft Genome of the Globally Widespread and Ivasive Argentine Ant ("A total of 7.184 genes (45%) were unique til L. Humile relative to these three ofther species," 2)
17, Tautz og Domazet-Loso, "The Evolutionary Origin of Orphan Genes"; "Rhizome of Life, Catastrophes, Sequence Exchanges, Gene Creations, and Giant virures"
18. Lyell, Principles of Geology
19. Se Pray og Zhaurova, "Barbara McClintock and the Discovery of Jumping Genes (Transposons)
20. Long et al, "The Origin of the new Genes", s.867
21. Nurminsky et al, "Selective Sweep of a Newly Evolved Sperm-Specific Gene ind Drosophila," s.574
22. Chen et al, "Evolution of Antifreeze GlycoProtein Gene from a Trypsinogen Gene in Antarctic Notothenioid Fish," 3816
23. Courseauz og Nahon, "Birth of Two Chimeric Genes in the Hominidae Lineage."
24. Knowles og McLysaght, "Recent de Novo Origin of Human Protein-Coding Genes"
25. Wu, Irwin og Chang, "de Novo Origin of Human Protein-Coding Genes"
26. Guerzoni og McLysaght, "De Novo Origins of Human Genes"; Wu et al, "De Novo Origin of Human Protein-Coding Genes"
27. Siepel, "Darwinian Alcymi"
28. Siepel, "Darwinian Alcymi"
29. Siepel, "Darwinian Alcymi"
30. Siepel skriver (ibid): " Disse tilsynelatende de novo gen-opprinnelsene, reiste spørsmålet om hvordan evolusjon ved naturlig utvalg kan produsere funksjonelle gener fra ikke-protein-kodende DNA. .. Hvordan kunne alle disse brikkene faller på plass gjennom tilfeldige prosesser av mutasjoner, rekombinasjoner og nøytral drift?
31. Selvsagt kunne en argumentere for at de mutasjonelle prosessene som Long argumenterer for, kan sees som forklaring på hvordan opprinnelse til nye gener fra pre-eksisterende kasetter av genetisk informasjon.. osv. i en lang sekvens bakover. Dette synet impliserer at selv om livets opprinnelse er et mystium, så kan prosessene som Long refererer til gjøre rede for informasjonsøkningen underveis. Likevel gjør ikke dette synet rede for hvordan skyfling av eksisterende informasjons-kasetter, genererer det spesifikke arrangementet av tegnene som utgjør innholdet i kasettene.
32. Zhang et al, "Adaptive Evolution of a Duplicated PanCreatic Ribonuclease Gene in a Leaf-Eating Monkey." Genene som koder for disse to proteinene skiller seg ved 12 nukleotider i sekvensen deres. Ut fra dette framsettes hypotese om en evolusjonær fordel for aper med det siste genet, og formodningen om et felles opphavs-gen (RNASE1 til RNASE1B) over tid.
36. Long et al, "The Origin of the new Genes", s.866
38. F.eks. A. A. Parikesit et al bemerker at "Selv om det ekisterer en statistisk siginfikant korrelasjon mellom protein-domene grenseområder og ekson grenseområder, så er omtrent 2/3 av de kommenterte protein-domene grenseområdene forstyrret med minst ett inron, og i gjennomsnitt inneholder et domene 3-4 introner"
39. Gauger, "Why Proteins Aren\t easily Recombined"
42. Nobuyusa et al, "Principles for Designing Ideal Protein Structures"; Marshall, "Proteins Made to order". Forskerne vedgår at det sannsynligvis er en 'trade-off' mellom funksjonalitet og stabilitet i naturlige proteiner. Det har foreløbig vist seg vanskelig å designe et protein som er både funksjonelt og stabilt i sin 3-dimensjonale form, fra scratch av..
43. Altamirano et al. "Directed Evolution of New Catalytic Activity Using the Alpha/beta/Barrel Scaffold." Se også Altamirano et al. "Retraction: directed evolution of New Catalytic Acitivity Using the Alpha/beta/Barrel Scaffold.
44. Gauger, "Why Proteins Aren\t easily Recombined"
Stoffutvalg og bilder ved Asbjørn E. Lund
.
 Tenk på ett gen
Tenk på ett gen Tenk på ett gen
Tenk på ett gen Ved nærmere undersøkelse viser ingen av disse artiklene hvordan mutasjoner og naturlig seleksjon, virkelig kunne finne nye gener, eller proteiner med 3-dimensjonal folding -i første omgang. Heller ikke viser de hvordan det kunne skje på tilgjengelig tid, verken i livets historie eller i forhold til kambrium. Artiklene formoder eksistensen av betydelige pre-eksisterende genetisk informasjon, og foreslår så ulike mekanismer som sakte kunne ha utviklet, eller slått sammen disse genene til større sammensetninger. I beste fall sporer disse artiklene historien til pre-eksistente gener, heller enn å forklare opphavet til de opprinnelige genene.
Ved nærmere undersøkelse viser ingen av disse artiklene hvordan mutasjoner og naturlig seleksjon, virkelig kunne finne nye gener, eller proteiner med 3-dimensjonal folding -i første omgang. Heller ikke viser de hvordan det kunne skje på tilgjengelig tid, verken i livets historie eller i forhold til kambrium. Artiklene formoder eksistensen av betydelige pre-eksisterende genetisk informasjon, og foreslår så ulike mekanismer som sakte kunne ha utviklet, eller slått sammen disse genene til større sammensetninger. I beste fall sporer disse artiklene historien til pre-eksistente gener, heller enn å forklare opphavet til de opprinnelige genene.  i) Like gensekvenser kan ha utviklet seg uavhengig i to parallelle utviklings-linjer, som starter med to ulike gener ('konvergent evolusjon'). Det er mange slike eks. nå (13). F.eks. benytter både flaggermus og hvaler, samme type ekkolokalisering, inklusive lignende gener og proteiner for dette. Dette har fått evolusjonsbiologer til å foreslå at de begge stammer fra en felles stamfar, som ikke opprinnelig hadde dette systemet. (14)
i) Like gensekvenser kan ha utviklet seg uavhengig i to parallelle utviklings-linjer, som starter med to ulike gener ('konvergent evolusjon'). Det er mange slike eks. nå (13). F.eks. benytter både flaggermus og hvaler, samme type ekkolokalisering, inklusive lignende gener og proteiner for dette. Dette har fått evolusjonsbiologer til å foreslå at de begge stammer fra en felles stamfar, som ikke opprinnelig hadde dette systemet. (14) Orfan gener
Orfan gener På dette viset knyttet Darwin til vitenskaps-prinsippet som en av hans vitenskapelige rolle-modeller, geologen Charlew Lyell, benyttet. Lyell insisterte på at gode forklaringer for opprinnelsen til geologiske trekk, skulle siterer 'årsaker som er i virksomhet nå, -årsaker som er kjent fra nåværende erfaring å ha kapasitet til å framskaffe den søkte effekten' (18). Hvordan er det med scenariene benyttet i Longs artikkel: tilfredsstiller de disse kriteriene? Dupliserings mutasjoner og andre typer tilfeldige mutasjoner, er sammen med naturlig seleksjon, klart årsaker som er i virksomhet nå. Men har disse prosessene demonstrert evnen til å produsere hva det spørres etter, nemlig ny genetisk informasjon som er nødvendig for strukturelle innovasjoner i livets historie? Det er flere gode grunnet til å mene at de ikke har gjort det.
På dette viset knyttet Darwin til vitenskaps-prinsippet som en av hans vitenskapelige rolle-modeller, geologen Charlew Lyell, benyttet. Lyell insisterte på at gode forklaringer for opprinnelsen til geologiske trekk, skulle siterer 'årsaker som er i virksomhet nå, -årsaker som er kjent fra nåværende erfaring å ha kapasitet til å framskaffe den søkte effekten' (18). Hvordan er det med scenariene benyttet i Longs artikkel: tilfredsstiller de disse kriteriene? Dupliserings mutasjoner og andre typer tilfeldige mutasjoner, er sammen med naturlig seleksjon, klart årsaker som er i virksomhet nå. Men har disse prosessene demonstrert evnen til å produsere hva det spørres etter, nemlig ny genetisk informasjon som er nødvendig for strukturelle innovasjoner i livets historie? Det er flere gode grunnet til å mene at de ikke har gjort det. Evolusjon av intet ('ex nihilo')?
Evolusjon av intet ('ex nihilo')? Forfatteren forutsetter at pre-adapsjon kan ha spilt en rolle. Men han tilføyer ingen ytterligere forklaring, siden det bare innebærer når (før seleksjon spilte en rolle) og hvor (ikke-protein kodende DNA), ikke hvordan de aktuelle genene oppsto. Evolusjonære biologer benytter typisk termen 'de novo opprinnelse' for å ha noe å henføre til, når de ikke kan forklare økning i genetisk informasjon. Det henfører ikke til noen kjent mutasjons-prosess. Så enten 'tigger' forklaringene spørsmål om opprinnelig informasjon, eller så foretar de uforklarlige hopp, evolusjonær skapelse 'ex nihilo'. Dermed forklarer ikke scenariene i Longs artikkel opprinnelsen til funksjonell spesifisert informasjon, verken i gener eller seksjoner av gener. Det ville kreve en årsak som kunne løse det kombinatoriske-søke spørsmålet i forrige kapittel. Men ingen av artiklene i Longs artikkel, verken adresserer eller demonstrerer matematisk troverdighet i mekanismene de henviser til. En kan ikke løse et problem eller imøtegå ett argument ved å unnlate å adressere det. (31) {Vi har tidligere presentert en ikke-tilfeldig evolusjonær hypotese i slike situasjoner.}
Forfatteren forutsetter at pre-adapsjon kan ha spilt en rolle. Men han tilføyer ingen ytterligere forklaring, siden det bare innebærer når (før seleksjon spilte en rolle) og hvor (ikke-protein kodende DNA), ikke hvordan de aktuelle genene oppsto. Evolusjonære biologer benytter typisk termen 'de novo opprinnelse' for å ha noe å henføre til, når de ikke kan forklare økning i genetisk informasjon. Det henfører ikke til noen kjent mutasjons-prosess. Så enten 'tigger' forklaringene spørsmål om opprinnelig informasjon, eller så foretar de uforklarlige hopp, evolusjonær skapelse 'ex nihilo'. Dermed forklarer ikke scenariene i Longs artikkel opprinnelsen til funksjonell spesifisert informasjon, verken i gener eller seksjoner av gener. Det ville kreve en årsak som kunne løse det kombinatoriske-søke spørsmålet i forrige kapittel. Men ingen av artiklene i Longs artikkel, verken adresserer eller demonstrerer matematisk troverdighet i mekanismene de henviser til. En kan ikke løse et problem eller imøtegå ett argument ved å unnlate å adressere det. (31) {Vi har tidligere presentert en ikke-tilfeldig evolusjonær hypotese i slike situasjoner.} Å forklare opphavet til strukturell innovasjon, innebærer mer enn bare å sammenligne ulike versjoner av samme gener, og forsøke spore et opphav til disse endringene. Det innebærer å finne forklaring til nytt genetisk materiale, som danner nye tredimensjonale protein-foldinger, slik at større strukturelle innovasjoner kan inntreffe. Så selv om disse scenariene i og for seg er greie nok, er de ikke relevante for å forklare opprinnelse til ny strukturell innovasjon, som forekommer i kambrium-perioden, -eller i noen andre begivenheter i livets historie.
Å forklare opphavet til strukturell innovasjon, innebærer mer enn bare å sammenligne ulike versjoner av samme gener, og forsøke spore et opphav til disse endringene. Det innebærer å finne forklaring til nytt genetisk materiale, som danner nye tredimensjonale protein-foldinger, slik at større strukturelle innovasjoner kan inntreffe. Så selv om disse scenariene i og for seg er greie nok, er de ikke relevante for å forklare opprinnelse til ny strukturell innovasjon, som forekommer i kambrium-perioden, -eller i noen andre begivenheter i livets historie. iii) En tredje mangel på samsvar finner vi i begrepet 'side-kjeder' (side chains). Alle de 20 protein-formende aminosyrene har sitt eget rammeverk (bakcbone-'ryggrad'), som kan kalles for en 'side-kjede'. Disse stikker ut i nesten rett vinkel fra rammeverket. Samhandlinger mellom slike 'side-kjeder' avgjør hvorvidt det vil bygges opp større, stabile 3-dimensjonale foldinger (39). Siden elementene i mindre, sekundære protein-enheter er omgitt av 'side-kjeder', så kan de ikke kombineres om ikke aktuelle side-kjeder er komplementære. Selv trente vitenskapsfolk, som har jobbet med proteiner i årevis, kan ha problemer med å designe sekvenser som kan gi stabile protein-foldinger (42). Manglende interaksjon fra side-kjedene (43) forhindrer det. Som molekylær-biolog Ann Gauger uttrykte det: " Således er alpha-helikser og 'beta-svøp' sekvens-avhengige strukturelle elementer av protein-foldinger. En kan ikke bytte dem rundt, som med Lego-brikker." (44) {Forskjellen er at mens Lego-fabrikanten driver utstrakt hemmelighold, over noe som relativt enkelt kan forstås, så har 'livets fabrikant' lagt koden åpent fram, men selv de skarpeste hoder har problemer med å forstå den -oversetters kommentar.}
iii) En tredje mangel på samsvar finner vi i begrepet 'side-kjeder' (side chains). Alle de 20 protein-formende aminosyrene har sitt eget rammeverk (bakcbone-'ryggrad'), som kan kalles for en 'side-kjede'. Disse stikker ut i nesten rett vinkel fra rammeverket. Samhandlinger mellom slike 'side-kjeder' avgjør hvorvidt det vil bygges opp større, stabile 3-dimensjonale foldinger (39). Siden elementene i mindre, sekundære protein-enheter er omgitt av 'side-kjeder', så kan de ikke kombineres om ikke aktuelle side-kjeder er komplementære. Selv trente vitenskapsfolk, som har jobbet med proteiner i årevis, kan ha problemer med å designe sekvenser som kan gi stabile protein-foldinger (42). Manglende interaksjon fra side-kjedene (43) forhindrer det. Som molekylær-biolog Ann Gauger uttrykte det: " Således er alpha-helikser og 'beta-svøp' sekvens-avhengige strukturelle elementer av protein-foldinger. En kan ikke bytte dem rundt, som med Lego-brikker." (44) {Forskjellen er at mens Lego-fabrikanten driver utstrakt hemmelighold, over noe som relativt enkelt kan forstås, så har 'livets fabrikant' lagt koden åpent fram, men selv de skarpeste hoder har problemer med å forstå den -oversetters kommentar.}